北京大学邹鹏团队开发定量蛋白质组学方法prox-SILAC揭示亚细胞区域内蛋白质动态变化
真核细胞具有纷繁复杂的三维空间结构。作为生命活动的主要执行者,蛋白质在多个亚细胞空间内合成、穿梭、代谢,并在特定的区域内发挥生物学功能。解析蛋白质的局部代谢和转运规律,以及其在不同生理状态下的变化,对于我们深刻理解生命过程的分子机制具有重要意义。尽管目前已有一些在全细胞水平表征蛋白质代谢速率的方法,如生物正交非天然氨基酸标记(BioOrthogonal Non-Canonical Amino acid Tagging,BONCAT)[1]及稳定同位素标记技术(pulse-SILAC)[2,3],但这些技术往往缺乏亚细胞水平上空间分辨率,难以深度揭示亚细胞区域内蛋白质组的动态变化规律。近年来,以生物素连接酶(BioID[4]/TurboID[5])和过氧化物酶(APEX2[6,7]/HRP[8])为代表的邻近标记技术在空间蛋白质组学领域得到了快速地发展,表现出优异的时空特异性和灵敏度。然而上述技术只能局限于亚细胞区域内蛋白质的丰度表征,而无法深层次的揭示蛋白质动态的代谢规律。
近日,北京大学邹鹏课题组在Nature Communications上发表了题为“Spatially resolved mapping of proteome turnover dynamics with subcellular precision”的研究。该研究结合过氧化物酶(APEX2/HRP)介导的邻近标记技术与pulse-SILAC定量蛋白质组学方法,开发了prox-SILAC技术,通过APEX2/HRP实现标记过程的空间特异性,凭借pulse-SILAC标记定义蛋白质代谢速率的时间尺度。利用prox-SLIAC技术,该研究实现对线粒体基质和内质网腔内蛋白质动态变化的精确鉴定,并揭示了内质网应激状态以及细胞分化过程中内质网腔内蛋白质代谢水平的差异变化。该技术的开发为亚细胞层面上蛋白质动态变化表征提供了有力工具。

该研究首先在HEK293T细胞线粒体基质中验证prox-SILAC方法的可行性(图1)。APEX2通过与细胞色素C氧化酶COX4的N端前导肽融合实现其在线粒体基质的表达。研究者首先利用重标SILAC培养基(Lys6,Arg8)对细胞进行4小时、8小时和12小时时间尺度的pulse-SILAC标记。随后在1分钟过氧化氢处理下,APEX2催化空间邻近的蛋白质与标记探针生物素-苯酚(biotin-phenol,BP)的共价偶联,实现生物素标记。富集后的生物素化蛋白质进一步通过LC-MS/MS予以鉴定和定量。研究者在这3个时间节点共鉴定并定量到183个蛋白质,其中162个(85%)此前存在线粒体定位注释(图1)。根据SILAC定量结果,研究者定义并计算了各蛋白质在一定时间内的代谢替换率(metabolic replacement fraction,MRF),用以衡量新合成蛋白质占蛋白质总量的比例。实验结果表明,不同生物学重复所测量得到的MRF值具有很好的相关性,表明了分析方法的稳定性和结果的可重复性。prox-SILAC标记揭示了线粒体基质蛋白质组整体代谢率的分布差异,特别是线粒体内膜上氧化呼吸链复合物及线粒体核糖体各亚基的动态变化,由此发现氧化呼吸链复合物I较其他复合物整体上具有更高的MRF值。与全细胞中pulse-SILAC方法相比,prox-SILAC同样揭示了一些氧化呼吸链亚基在线粒体中具有较全细胞中更高的MRF值。
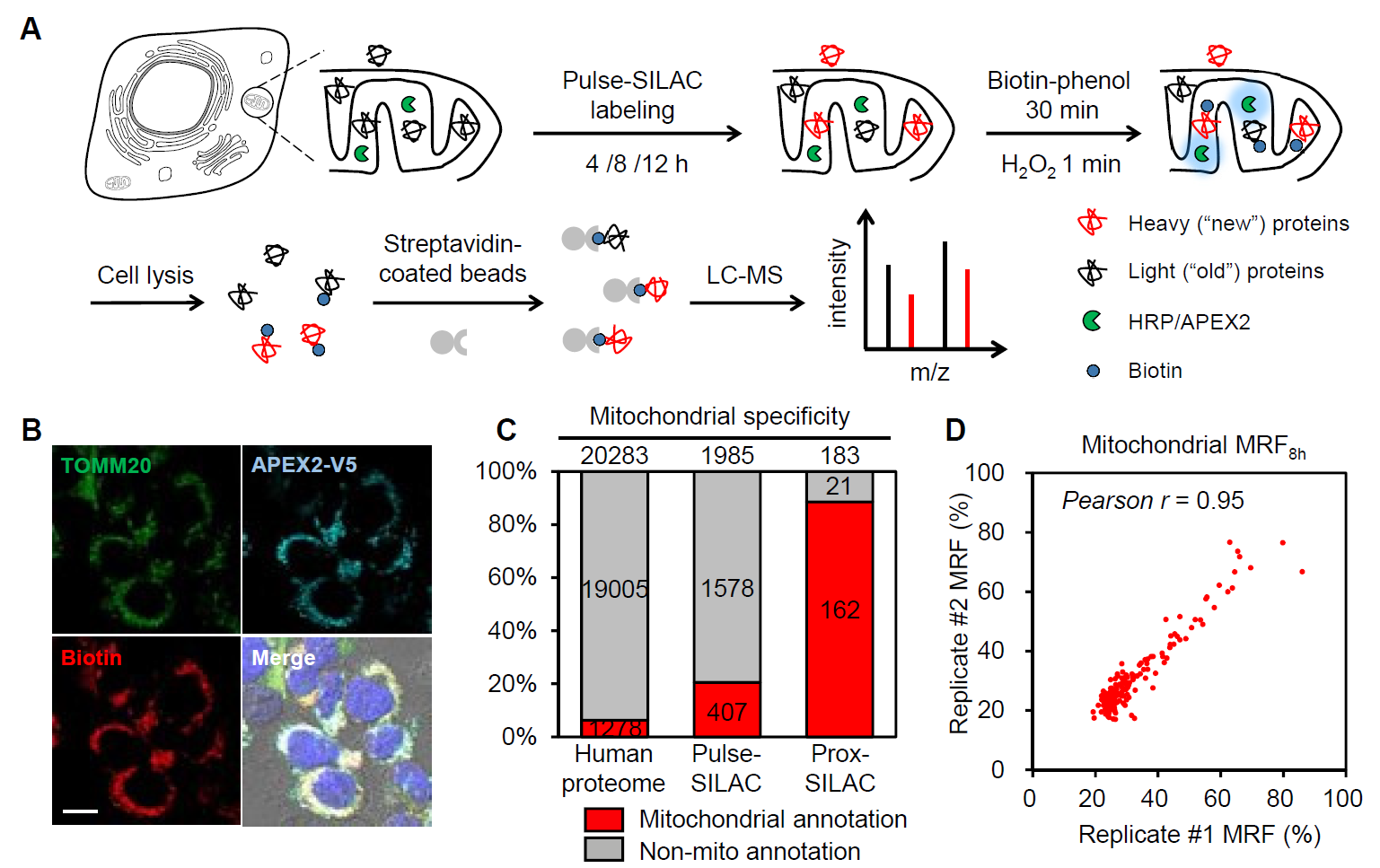
图1. prox-SILAC标记方法在线粒体基质中的应用
随后,研究者进一步利用prox-SILAC方法鉴定了内质网腔内蛋白质的动态变化,内质网腔内蛋白质的邻近标记由辣根过氧化物酶HRP所介导(图2)。在4小时、8小时、12小时的时间节点下,该研究同时鉴定到186个蛋白质,其中97%的蛋白质都存在分泌通路定位,ER驻留蛋白较后分泌通路蛋白整体具有更低的MRF值。与全细胞pulse-SILAC实验结果相比较,后分泌蛋白质同样在内质网腔内具有更高的新合成比例。研究者进一步对细胞表面膜蛋白质的代谢情况进行了测量,对比发现蛋白质的代谢水平与所处的亚细胞定位密切相关。如膜表面受体蛋白和离子泵蛋白TFRC、ATP1A1、IGF2R在内质网腔中具有显著更高的MRF值;而分子伴侣SERPINH1和PDIA6在内质网腔中的MRF值远小于其在细胞表面。由此可以看出,蛋白质在不同亚细胞区域内具有迥异的MRF值。这一方面反应了蛋白质合成后转运的时间轨迹,另一方面也可能涉及蛋白质存在多重定位和多重功能。
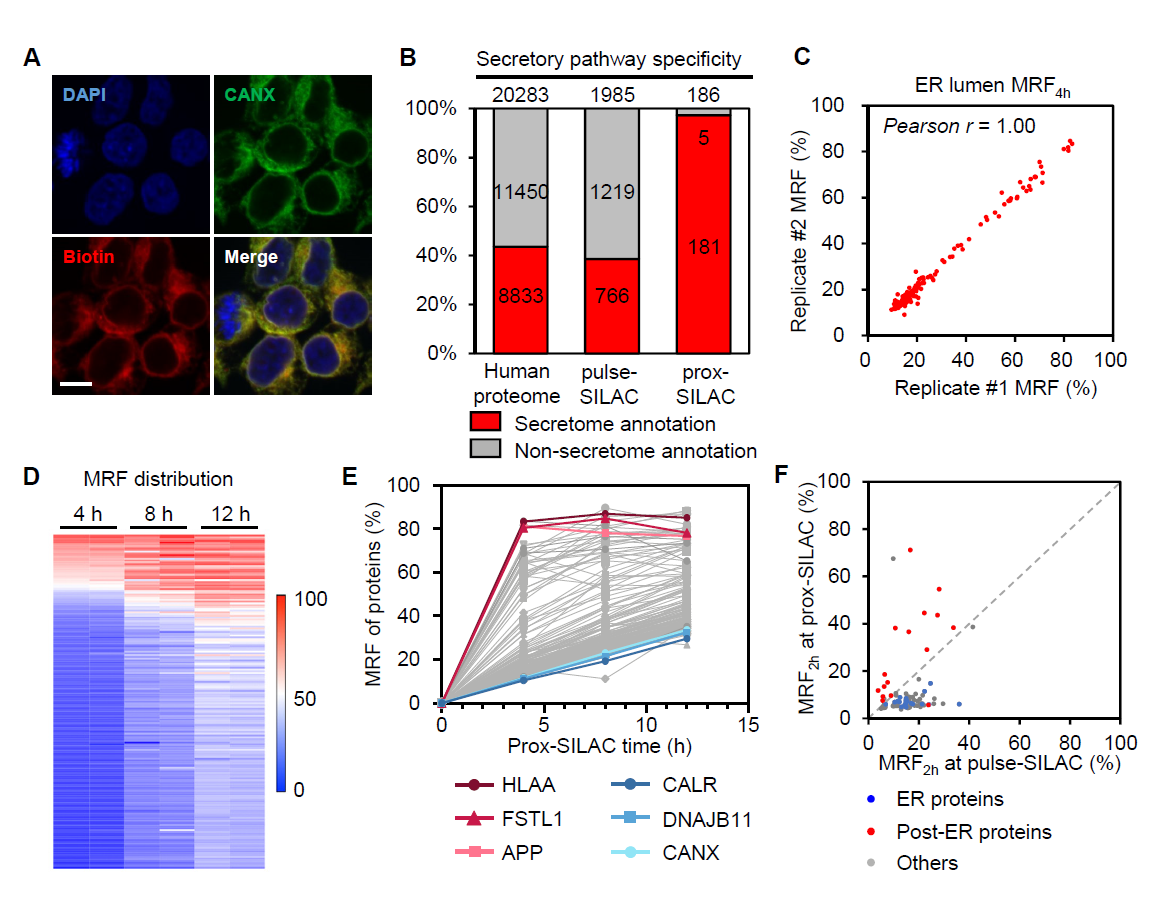
图2. prox-SILAC技术揭示内质网腔内蛋白质动态变化
相比于生理状态下,在病理状态或外界刺激干扰条件下对蛋白质稳态变化的研究对于理解疾病或药物作用机理具有更重要的意义。研究者分别考察了毒胡萝卜素刺激诱发内质网应激和细胞分化过程中的内质网腔内的蛋白质代谢速率的变化。在内质网应激条件下,内质网腔内蛋白质代谢速率整体放缓,而该趋势无法通过全细胞pulse-SILAC实验所鉴定。此外本研究还鉴定到HSPA5、HSPA6、JAGN1和LMAN1等蛋白在,在毒胡萝卜素作用下代谢速率显著上调。而在反式维甲酸和脑源神经营养因子所诱导的SH-SY5Y细胞分化模型中,研究者分别在分化第0天、第7天和第10天进行prox-SILAC实验,考察内质网腔内蛋白质动态变化。研究者发现大部分内质网腔蛋白质在分化早期(第0天至第7天)代谢水平整体上调,而在分化后期(第7天至第10天)逐渐减缓。同样地,该变化趋势仍无法通过pulse-SILAC体现,凸显了亚细胞层面表征蛋白质动态变化的意义。
总结一下,该研究整合APEX标记技术与pulse-SILAC技术,开发了prox-SILAC方法,成功鉴定了线粒体和内质网腔内的蛋白质组动态变化,揭示了蛋白质具有亚细胞区域特异的代谢速率,为深刻解读蛋白质稳态与转运过程提供了重要化学生物学工具。值得注意的是,prox-SILAC标记在一定程度上受限于APEX2酶活性与标记偏好性,因此所鉴定到的蛋白质种类和定量结果可能存在一定偏差。
生命科学联合中心、北京大学化学与分子工程学院邹鹏研究员为本论文的通讯作者,已毕业博士研究生袁锋和李逸为本论文共同第一作者。该工作得到了国家自然科学基金委、科技部、北京分子科学国家研究中心、生物有机与分子工程教育部重点实验室、北京脑科学与类脑研究所的支持。
原文链接:https://doi.org/10.1038/s41467-023-42861-8
参考文献
ADDIN EN.REFLIST [1] Dieterich, D. C., Link, A. J., Graumann, J. et al. Selective identification of newly synthesized proteins in mammalian cells using bioorthogonal noncanonical amino acid tagging (BONCAT). Proc Natl Acad Sci U S A 103, 9482-9487 (2006).
[2] Snider, J., Wang, D., Bogenhagen, D. F. et al. Pulse SILAC Approaches to the Measurement of Cellular Dynamics. Adv Exp Med Biol 1140, 575-583 (2019).
[3] Boisvert, F. M., Ahmad, Y., Gierliński, M. et al. A quantitative spatial proteomics analysis of proteome turnover in human cells. Mol Cell Proteomics 11, M111.011429 (2012).
[4] Roux, K. J., Kim, D. I., Raida, M. et al. A promiscuous biotin ligase fusion protein identifies proximal and interacting proteins in mammalian cells. J Cell Biol 196, 801-810 (2012).
[5] Branon, T. C., Bosch, J. A., Sanchez, A. D. et al. Efficient proximity labeling in living cells and organisms with TurboID. Nat Biotechnol 36, 880-887 (2018).
[6] Martell, J. D., Deerinck, T. J., Sancak, Y. et al. Engineered ascorbate peroxidase as a genetically encoded reporter for electron microscopy. Nat Biotechnol 30, 1143-1148 (2012).
[7] Lam, S. S., Martell, J. D., Kamer, K. J. et al. Directed evolution of APEX2 for electron microscopy and proximity labeling. Nat Methods 12, 51-54 (2015).
[8] Kim, D. I. & Roux, K. J. Filling the Void: Proximity-Based Labeling of Proteins in Living Cells. Trends Cell Biol 26, 804-817 (2016).
