邹鹏课题组利用光催化邻近标记技术揭示应激颗粒转录组动态变化
应激颗粒是细胞在受到外界刺激条件时应激产生的一种无膜细胞器,不仅可以在几十分钟内迅速组装形成,还能够在刺激条件去除后逐渐解体,展现出了高度的动态特性。应激颗粒中不仅含有大量RNA分子,还存在多种参与蛋白质翻译的因子,这些组份对于维持应激颗粒结构的稳定和调控蛋白质翻译过程至关重要1。尽管目前关于应激颗粒中的RNA成分问题已有不少研究,但对于其组装/解聚过程中RNA组成的变化仍然知之甚少。例如,基于成像的技术能够直接观测不同转录本在应激颗粒中的定位,以及可以实现在活细胞中监测转录本在不同颗粒之间的交换与翻译状态。然而,成像技术受限于较低的通量,并且需要已知的序列信息设计引物或构建报告分子,难以对应激颗粒中的RNA组成进行全局研究2-3。基于抗体的亲和沉淀分离应激颗粒并结合高通量测序技术能够提供应激颗粒的转录组信息4。然而在实际操作中,复杂的分离纯化步骤很容易造成组分丢失或引入污染,从而导致转录组数据含有较高的假阴性与假阳性。同时,这种分离方法无法应用于研究应激颗粒的组装与解聚动态过程。
近日,生命中心、北京大学化学与分子工程学院邹鹏课题组在Nature Communications上发表题为“Profiling stress-triggered RNA condensation with photocatalytic proximity labeling”的论文,利用遗传靶向的光催化邻近标记技术CAP-seq首次解析了应激颗粒转录组的动态变化5。在蓝光激发下,蛋白质光催化剂miniSOG能够介导对邻近RNA分子上鸟嘌呤的氧化损伤,同时将一个带有生物正交官能团的氨基探针共价交联到损伤碱基,从而实现在~20纳米的空间尺度下对亚细胞区域中RNA的标记。该工作首先将miniSOG与应激颗粒的核心组份蛋白质G3BP1融合表达在人胚胎肾细胞HEK293T中,利用亚砷酸钠氧化胁迫或山梨糖醇渗透压胁迫两种方式诱导应激颗粒的产生,并开展CAP-seq标记其中的RNA组分。在两种刺激条件下分别获得457和822个在应激颗粒处富集的转录本,并通过后续的单分子荧光成像实验对其中部分新发现的RNA进行了验证。由此也发现了应激颗粒的RNA组分在不同刺激条件下存在143个共有的转录本成分,这为深入解析应激颗粒的组装机制提供了重要线索。
更进一步,该工作利用CAP-seq能够直接标记RNA的优势首次解析了应激颗粒在动态变化下的转录组组成,包括在无刺激的本底水平下存在的应激颗粒相关作用网络中的RNA组成,以及在亚砷酸钠刺激撤去后应激颗粒解聚过程中基于G3BP1蛋白的核心组分中RNA成的变化。该工作发现,在刺激下转录本上CDS与3’ UTR序列上丰富的AU组成能够促进RNA靶向至应激颗粒。在应激颗粒解聚过程中,具有更长3’ UTR且AU含量高的转录本倾向于保留在应激颗粒中,同时无刺激下应激颗粒核心组分中的mRNA具有较高m6A修饰水平,并且能够促进mRNA在应激颗粒解聚初期仍定位在应激颗粒的核心结构中。此外,该工作发现在应激颗粒基本解聚后,其核心组分的RNA组成更加类似于成熟应激颗粒中的RNA组分特征。对以上数据一种可能的解读是:应激颗粒在解聚后,虽然在光学成像分辨率下看起来已经复原为刺激前的静息状态,然而其核心组分与刺激前相比有所改变。这种局部组分的重塑在一定程度上可以理解为细胞的记忆效应,即短暂的外界刺激可能在细胞中造成长期的影响,从而产生细胞的“应激记忆”。“应激记忆”模型从另一个视角为我们诠释了应激颗粒的生物学功能,也拓展了我们对于细胞内发生液-液相分离过程的认识。
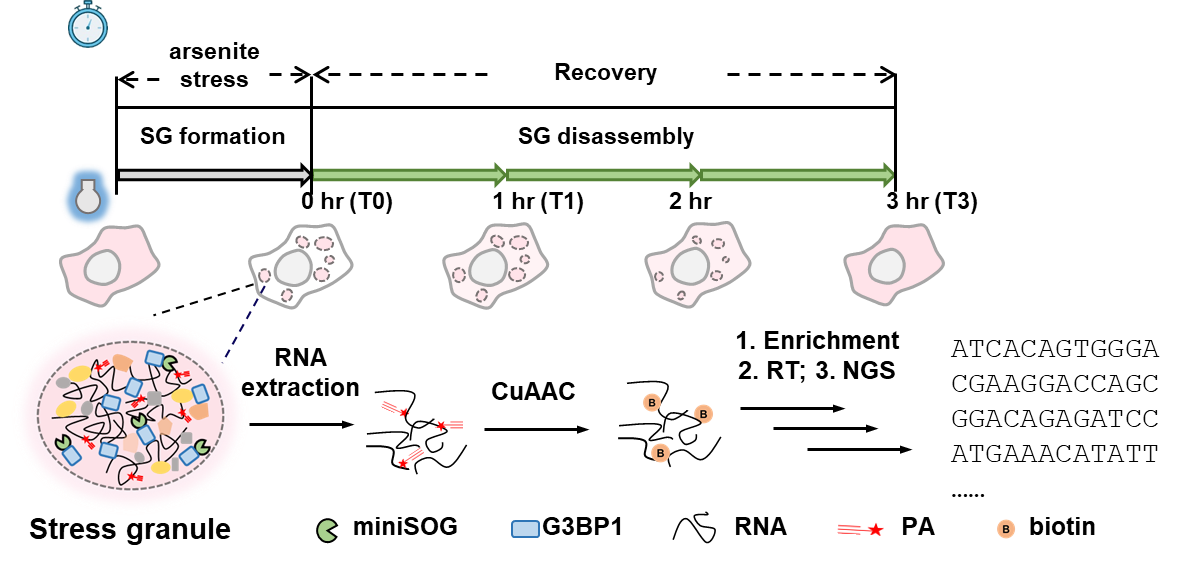
图1 基于CAP-seq解析不同条件下的应激颗粒转录组组分。
北京大学化学与分子工程学院、生命科学联合中心邹鹏研究员为该论文的通讯作者,博士研究生任子琪与课题组已毕业的唐微博士为该论文的共同第一作者。该工作得到了国家自然科学基金委、科技部、北京分子科学国家研究中心、生物有机与分子工程教育部重点实验室、生命科学联合中心、北京脑科学与类脑研究所的支持。
参考文献
1. Protter, D. S. W. & Parker, R. Principles and properties of stress granules. Trends Cell Biol. 26, 668-679 (2016).
2. Mateju, D. et al. Single-molecule imaging reveals translation of mRNAs localized to stress granules. Cell 183, 1-12 (2020).
3. Wilbertz, J. H. et al. Single-molecule imaging of mRNA localization and regulation during the integrated stress response. Mol. Cell 73, 946-958.e947 (2019).
4. Khong, A. et al. The stress granule transcriptome reveals principles of mRNA accumulation in stress granules. Mol. Cell 68, 808-820.e805 (2017).
5. Wang, P. et al. Mapping spatial transcriptome with light-activated proximity-dependent RNA labeling. Nat. Chem. Biol. 15, 1110-1119 (2019).
