周帆课题组与合作者构建和鉴定非人灵长类类胚胎模型
胚胎着床至原肠运动是发育的关键时期,也是临床早期流产的高发期。受限于伦理争议和样本来源,相比于模式动物,灵长类围着床后发育的相关研究明显滞后。基于胚胎干细胞组装的类胚胎模型为灵长类胚胎发育研究提供了新的途径。目前小鼠胚胎干细胞来源的类胚胎能体外培养到神经发生和器官发生阶段【1-3】,人胚胎干细胞诱导获得的类囊胚也可以模拟囊胚阶段的形态和分子特征【4-7】。非人灵长类具有与人相近的生殖发育和胚胎发育特点,是研究生殖和胚胎发育的理想动物模型。然而,适用于非人灵长类胚胎研究的类胚胎模型至今未见报道。
2023年4月6日,生命科学联合中心周帆课题组、中国科学院脑科学与智能技术卓越创新中心刘真课题组和孙强课题组合作在Cell Stem Cell期刊在线发表了题为Cynomolgus monkey embryo model captures gastrulation and early pregnancy的研究论文。该研究利用胚胎干细胞构建了含有主要谱系和结构特征的食蟹猴类囊胚,体外培养后可诱导其产生原肠胚的形态与细胞群体,体内移植后可检测到早期妊娠囊等早孕特征。

图1. 结合干细胞体外重构培养和单细胞转录组分析,构建与鉴定食蟹猴类胚胎
首先,研究人员利用非人灵长类胚胎干细胞进行食蟹猴类囊胚的体外构建。通过比较不同培养状态的食蟹猴胚胎干细胞并进一步优化体外诱导体系和方法,最终确认了4CL原始态(Naïve)胚胎干细胞体系与HDM-mTDM两步法诱导,能够高效获得和食蟹猴生理囊胚形态上接近的类囊胚(图1A)。进一步的单细胞转录组测序分析、体外分离与干细胞建系等实验,提示新构建的食蟹猴类囊胚具有与生理囊胚相似的细胞谱系以及转录组特征(图1B)。
接下来,为鉴定其体外发育潜能,作者对食蟹猴类囊胚进行了长时程体外延迟培养。在体外培养至D17,类囊胚发育为圆盘样类胚胎,并包含羊膜腔、卵黄囊腔等特征性结构,且部分形态与之前报道的人类CS7(E16-19)时期的体内原肠胚相似【8】(图1C)。结合转录组测序数据挖掘和对比分析,作者发现D17食蟹猴类胚胎具有三胚层类似的细胞群体(图1D)。这些结果提示D17食蟹猴类胚胎无论从形态上,还是细胞谱系组成上,都与人类CS7(E16-19)胚胎具有相似特征,提示新构建的类囊胚具有一定着床后发育潜能。
最后,作者将食蟹猴类囊胚进行同种体内移植,结果显示部分受体在移植后能检测到明显的着床点和早期妊娠囊,同时伴随受体血清中绒毛膜促性腺激素和孕激素水平升高,暗示能诱发一定的早孕反应特征(图2)。
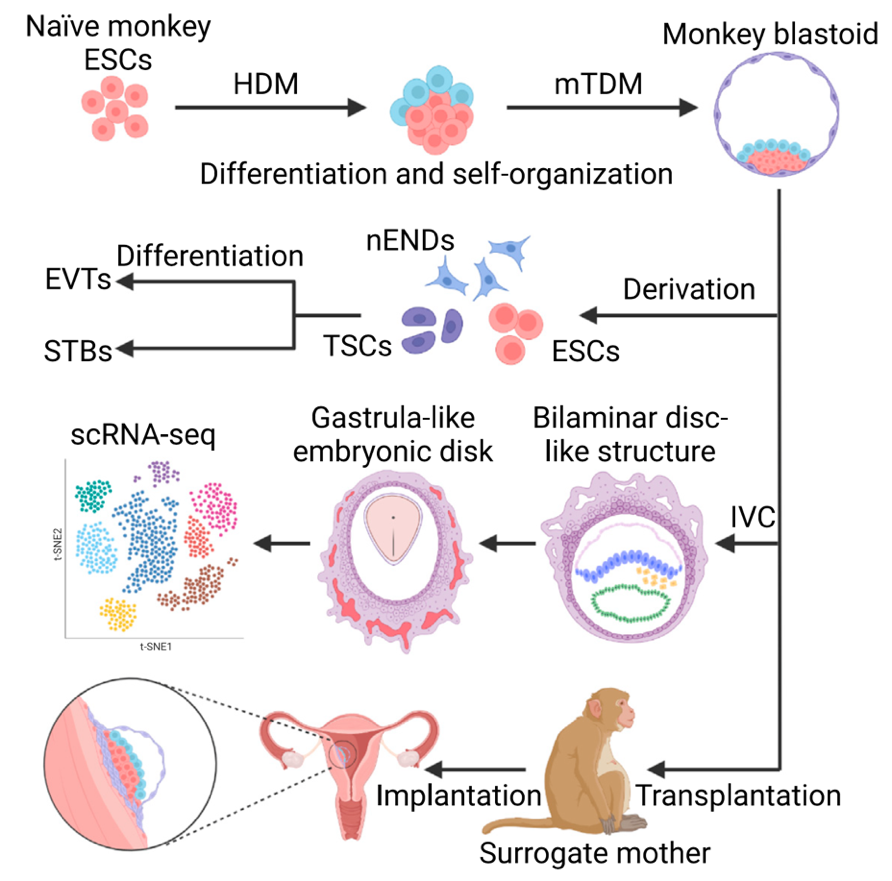
图2. 非人灵长类类囊胚体外重构和体内外发育潜能研究模式图
综上,该研究利用胚胎干细胞体外重构了非人灵长类类囊胚,并对其体内外发育潜能进行深入的探究和评估。将非人灵长类的类囊胚体外培养至类原肠胚时期,为进一步优化灵长类类囊胚的体外重构策略,以及深入理解灵长类的胚胎发育过程提供了有价值的研究模型。
中科院脑智卓越中心刘真研究员、生命中心PI周帆、中科院脑智卓越中心孙强研究员为该论文的共同通讯作者。中科院脑智卓越中心博士后李杰、周帆课题组科研助理朱庆元、中科院脑智卓越中心与西北农林科技大学联合培养博士生曹静、周帆课题组2021级博士生刘莹、以及非人灵长类研究平台实验师陆勇为该论文并列第一作者。周帆课题组2021级博士生李倩等为本研究提供了重要帮助。
原文链接: https://doi.org/10.1016/j.stem.2023.03.009
参考文献
[1] Lau, K.Y.C., Rubinstein, H., Gantner, C.W., Hadas, R., Amadei, G., Stelzer, Y., and Zernicka-Goetz, M. (2022). Mouse embryo model derived exclusively from embryonic stem cells undergoes neurulation and heart development. Cell Stem Cell 29, 1445-1458 e1448. 10.1016/j.stem.2022.08.013.
[2] Tarazi, S., Aguilera-Castrejon, A., Joubran, C., Ghanem, N., Ashouokhi, S., Roncato, F., Wildschutz, E., Haddad, M., Oldak, B., Gomez-Cesar, E., et al. (2022). Post-Gastrulation Synthetic Embryos Generated Ex Utero from Mouse Naïve ESCs. Cell. 10.1016/j.cell.2022.07.028.
[3] Amadei, G., Handford, C.E., Qiu, C., De Jonghe, J., Greenfeld, H., Tran, M., Martin, B.K., Chen, D.Y., Aguilera-Castrejon, A., Hanna, J.H., et al. (2022). Synthetic embryos complete gastrulation to neurulation and organogenesis. Nature. 10.1038/s41586-022-05246-3.
[4] Yu, L., Wei, Y., Duan, J., Schmitz, D.A., Sakurai, M., Wang, L., Wang, K., Zhao, S., Hon, G.C., and Wu, J. (2021). Blastocyst-like structures generated from human pluripotent stem cells. Nature. 10.1038/s41586-021-03356-y.
[5] Liu, X., Tan, J.P., Schroder, J., Aberkane, A., Ouyang, J.F., Mohenska, M., Lim, S.M., Sun, Y.B.Y., Chen, J., Sun, G., et al. (2021). Modelling human blastocysts by reprogramming fibroblasts into iBlastoids. Nature. 10.1038/s41586-021-03372-y.
[6] Kagawa, H., Javali, A., Khoei, H.H., Sommer, T.M., Sestini, G., Novatchkova, M., Scholte Op Reimer, Y., Castel, G., Bruneau, A., Maenhoudt, N., et al. (2021). Human blastoids model blastocyst development and implantation. Nature. 10.1038/s41586-021-04267-8.
[7] Yanagida, A., Spindlow, D., Nichols, J., Dattani, A., Smith, A., and Guo, G. (2021). Naive stem cell blastocyst model captures human embryo lineage segregation. Cell Stem Cell. 10.1016/j.stem.2021.04.031.
[8] Tyser, R.C.V., Mahammadov, E., Nakanoh, S., Vallier, L., Scialdone, A., and Srinivas, S. (2021). Single-cell transcriptomic characterization of a gastrulating human embryo. Nature 600, 285-289. 10.1038/s41586-021-04158-y.
