DNA甲基化在基因表达、基因印记和X染色体失活等过程中发挥着重要的调控功能,其异常将导致胚胎发育缺陷、不育及多种人类疾病。作为细胞表观记忆机制的重要组成部分,DNA甲基化在体细胞分裂中能够稳定遗传。然而,在哺乳动物配子发生和早期胚胎发育过程中,DNA甲基化经历了广泛而剧烈的重编程,以完成亲代-子代转变和发育时钟的重置。在哺乳动物的整个生命周期中,DNA甲基化经历了三次大规模的重新建立过程,分别发生于精子发生、卵子发生和胚胎着床之后。其中,精子发生和卵子发生阶段的DNA甲基化建立分别受组蛋白修饰H3K36me2和H3K36me3严格调控[1, 2],但胚胎着床后DNA甲基化重建的分子机制仍不清楚。
2025年11月11日,生命中心/清华大学生命学院颉伟、山东大学妇儿与生殖健康研究院卢绪坤和复旦大学生殖与发育研究院张宇团队合作,在Nature Cell Biology发表题为“H3K36me2重编程谱系特异性调控着床后DNA甲基化从头建立”(Reprogramming of H3K36me2 guides lineage-specific post-implantation de novo DNA methylation)的研究论文。该研究通过系统检测小鼠早期胚胎发育过程中H3K36me2的动态重编程,揭示了H3K36me2重编程协同DNMT3A/3B以谱系特异性的方式调控着床后DNA甲基化重建的功能和分子机制。
研究者首先系统绘制了小鼠卵母细胞、着床前和着床后胚胎中的H3K36me2动态变化图谱,发现卵母细胞转录影响H3K36me2与H3K36me3之间的相互转变。在转录沉默的卵母细胞中,H3K36me2更倾向于定位到基因体(gene body)区域。受精之后,父本H3K36me2快速丢失,而母本H3K36me2在8-细胞之后丢失。合子基因组激活(Zygotic genome activation,ZGA)之后,H3K36me2呈现“两步”建立过程:着床前胚胎H3K36me2首先在增强子逐步建立(Seeding),着床后遍布大部分基因组(失活的X染色体除外)(Spreading)。为了研究H3K36me2在着床后DNA甲基化重建中的功能和对胚胎发育的影响,研究者构建了H3K36me2甲基转移酶——NSD1酶活突变的小鼠模型。结果显示,H3K36me2缺失显著影响胚外组织中DNA甲基化的重建,但是对胚胎组织中DNA甲基化重建的影响较小。进一步机制研究表明,DNMT3A通过PWWP结构域严格识别和依赖H3K36me2/3(Accurate reader),而DNMT3B则可以不依赖H3K36me2/3进行DNA甲基化(Leaky reader)。因此,胚胎组织中高表达的DNMT3B在H3K36me2缺失的情况下仍可以保证全局性DNA甲基化的建立。但是,H3K36me2对于经常受到甲基化调控的启动子,包括生殖系特异性基因启动子的有效甲基化和沉默是必需的。研究还发现,富含发育关键基因的DMV(DNA methylation valley)区域能够通过PRC1/H2AK119ub1抑制H3K36me2,进而防止该区域被异常甲基化。
值得关注的是,在小鼠精子和卵子中,DNMT3B通常低表达甚至不表达,而DNMT3A是最重要的从头DNA甲基化酶,由于DNMT3A严格依赖H3K36me2/3,研究者认为这种分工可能保证了基因印记(一种控制两性繁殖的亲本差异化DNA甲基化现象)能够在精卵中准确建立。
综上所述,该研究揭示了H3K36me2在小鼠早期胚胎发育中的重编程规律,包括其两步建立过程,并确定了H3K36me2在着床后胚胎中谱系和位点特异性DNA甲基化重建及基因调控中的重要作用。此外,研究还解析了DNMT3A和DNMT3B在建立DNA甲基化时对H3K36me2/3的不同依赖性。该研究不仅加深了我们对不同生物学过程(精子发生、卵子发生和着床后胚胎发育)中DNA甲基化建立调控机制的理解,也将为不孕不育的诊断治疗和更精准表观组编辑工具的开发提供理论依据。
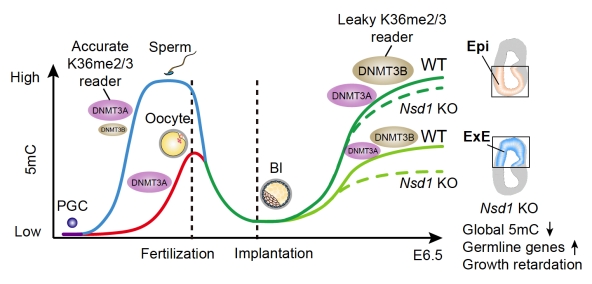
图1. H3K36me2协同DNMT3A/3B谱系特异性调控着床后DNA甲基化的重建
生命中心/清华大学颉伟教授、山东大学妇儿与生殖健康研究院卢绪坤副研究员(颉伟实验室原博后)和复旦大学生殖与发育研究院张宇青年研究员为本文共同通讯作者。卢绪坤副研究员和清华大学生命学院2023级博士生王利娟为本文共同第一作者。清华大学博士后刘伯峰、博士后胡晓瑜、2017级博士毕业生王正茂、科研助理刘玲、博士后于广、科研助理董丽君、2022级博士生孔凤、2017级博士毕业生范强在本研究中做出了重要贡献。本研究得到国家重点研发计划、国家自然科学基金、上海市自然科学基金、清华-北大生命科学联合中心、新基石基金的经费支持。同时也得到了清华大学实验动物中心、生物医学测试中心基因测序平台以及计算平台的大力协助和支持。
论文链接:https://www.nature.com/articles/s41556-025-01805-8
参考文献:
1. Shirane, K., et al., NSD1-deposited H3K36me2 directs de novo methylation in the mouse male germline and counteracts Polycomb-associated silencing. Nat Genet, 2020. 52(10): p. 1088-1098.
2. Xu, Q., et al., SETD2 regulates the maternal epigenome, genomic imprinting and embryonic development. Nat Genet, 2019. 51(5): p. 844-856.
